

自交不亲和(Self-incompatibility,SI)是开花植物在长期进化过程中形成的一种阻止自花授粉结实的现象。基于S-RNase的配子体自交不亲和(Gametophytic self-incompatibility,GSI)系统在双子叶植物较为常见,例如蔷薇科、茄科、车前科和芸香科植物中。目前,S-RNase基因在双子叶植物中的进化起源尚不清晰。
近日,我院赵涛教授/马锋旺教授课题组在Plant Physiology杂志在线发表题为S-RNase Evolution in Self-Incompatibility: Phylogenomic Insights into Synteny with Class I T2 RNase Genes的研究论文。

该研究利用130个代表性被子植物基因组,基于课题组前期开发的全基因组共线性网络分析方法(Zhao et al, Plant Cell, 2017; Zhao & Schranz, PNAS, 2019),从系统发育基因组学的角度,整合系统发育重建和基因组共线性信息,系统厘清了双子叶植物中S-RNase基因的系统发育关系与进化特点。前人研究报道S-RNase基因属于RNase T2基因家族的Class III分支,该研究进一步将Class III分支细化为四个亚类:Class III-A(包含已功能报道的S-RNase基因)、Class III-B、Class III-C和Class III-D,研究发现葫芦科物种中Class III-A的部分S-like RNase基因与已功能报道的S-RNase基因具有高度序列相似性,为其直系同源基因;共线性网络分析发现,葫芦科物种中Class III-A的部分S-like RNase基因与Class I的T2 RNase基因存在显著保守的共线性关系,进一步通过 Ks 及核型分析推测葫芦科Class III-A的部分S-like RNase基因(S-RNase的直系同源基因)与Class I的T2RNase基因共同起源于核心真双子叶植物共享的全基因组三倍化事件(γ-triplication)。
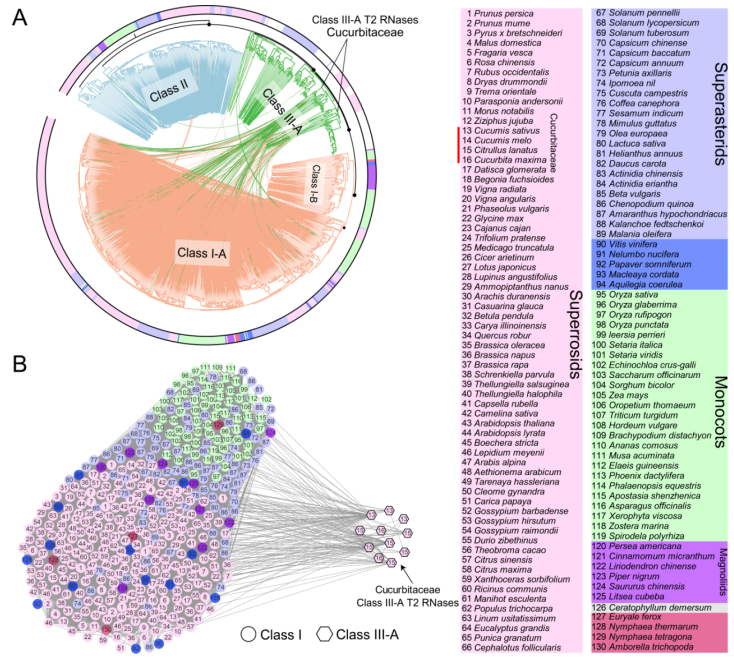
图1 RNase T2基因家族在130个代表性植物基因组中的共线性网络分析
图2 葫芦科Class III-AT2RNase基因是S-RNase的直系同源基因
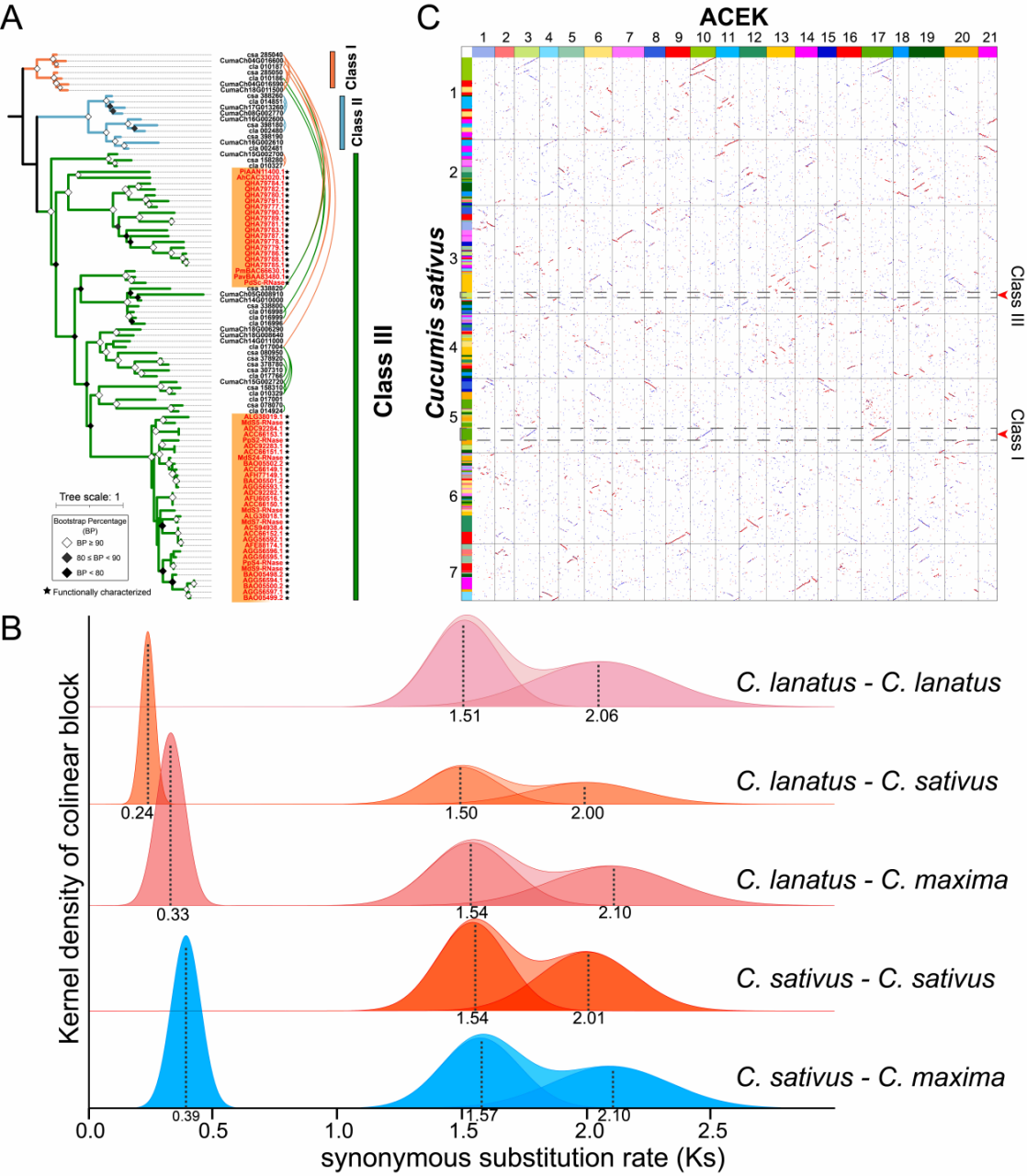
图3 葫芦科Class III-A与Class IT2 RNase基因的共线性、 Ks 及核型分析
此外,该研究还发现Class III-A中的部分S-RNase基因和S-like RNase基因在双子叶植物的不同谱系(蔷薇科、茄科和芸香科)中发生了特异性基因组位置转位现象,例如蔷薇科植物苹果族(苹果、梨、枇杷)与李属(桃、果梅、中国李)基因组中具有序列高度同源的S-RNase基因,但在基因组中的位置发生了特异性转位,此现象可能与苹果族植物所特有的全基因组加倍(WGD,Whole genome duplication)事件相关;且S-RNase基因附近的转座子元件活性显著增强。该研究系统揭示了双子叶植物S-RNase基因的进化起源,特异性基因转位与演化路径,研究结果为进一步的功能研究奠定了基础。
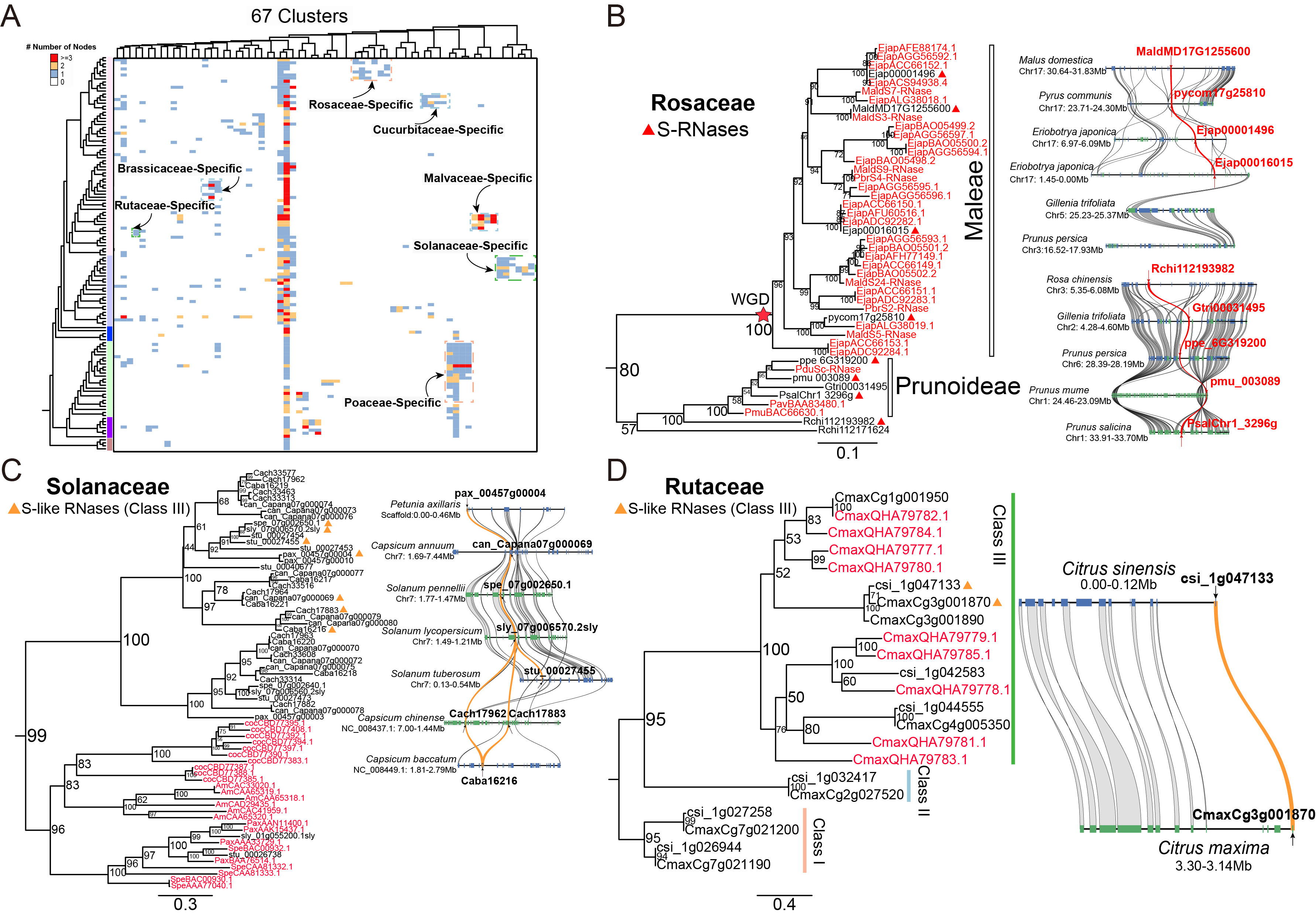
图4 S-RNase和S-like RNase基因的科属特异性基因组位置转位
我院在读博士研究生刘云霄为论文第一作者,赵涛教授、马锋旺教授和关长飞副教授为该论文的共同通讯作者,学院孙亚强副教授、南京农业大学薛佳宇副教授、华中农业大学
林宗成教授和荷兰瓦赫宁根大学Eric Schranz教授也参与了该项研究。该研究得到了国家重点研发(2023YFD1202400)、国家自然科学基金32102338)、中央高校基本科研业务费(2452021133)和西北农林科技大学高性能计算平台的支持。
论文链接:
https://doi.org/10.1093/plphys/kiaf072